Cell:泛修饰测序揭示核糖体表观遗传组的热调控机制
来源:BioArt 2025-12-01 14:20
这项研究通过Pan-Mod-seq、PANORAMA和电镜分析等技术,首次系统揭示rRNA修饰的动态性与环境适应性,为后续进一步理解rRNA表观遗传组在健康和疾病状态中的作用铺平道路。
核糖体是蛋白质翻译的工厂,由高度保守的rRNA和蛋白质构成。研究表明rRNA存在广泛的转录后修饰,且修饰常发生在功能中心,由小核RNA(small nucleolar RNAs,snoRNAs)(真菌,古细菌为snoRNA样RNA)催化介导【1,2】。rRNA修饰的缺失与rRNA前体加工破坏、异常翻译和机体适应性降低有关(详见BioArt报道:Mol Cell丨RNA指纹图谱揭示肿瘤起源和特性)【1】。一个关键问题是rRNA修饰多大程度是可变的。传统观点认为rRNA修饰是静态的;然而,最新研究指出一部分rRNA修饰处于动态变化【3,4】,例如嗜热古菌Thermococcus kodakarensis的数百个rRNA胞嘧啶发生温度诱导的乙酰化(ac4C)【5】。尽管如此,目前对rRNA动态修饰的程度、调节和功能意义依然有限。rRNA修饰的系统性研究一直面临三大挑战:多数方法只能检测单一修饰;通量低,难以并行处理多样本;许多修饰尚无可靠的测序检测方法。
近日,以色列魏茨曼科学研究所分子遗传学系Schraga Schwartz团队在Cell上发表了题为Pan-modification profiling facilitatesacross-evolutionary dissectionof the thermoregulated ribosomal epitranscriptome的研究文章。该研究开发高通量、多修饰和多样本并行检测的Pan-Mod-seq,系统揭示了rRNA修饰在热适应性中的核心作用,特别是m⁵C和ac⁴C的协同热稳定机制,不仅推动了表观转录组学方法的发展,也为理解生物在极端环境中的适应机制提供了全新视角。

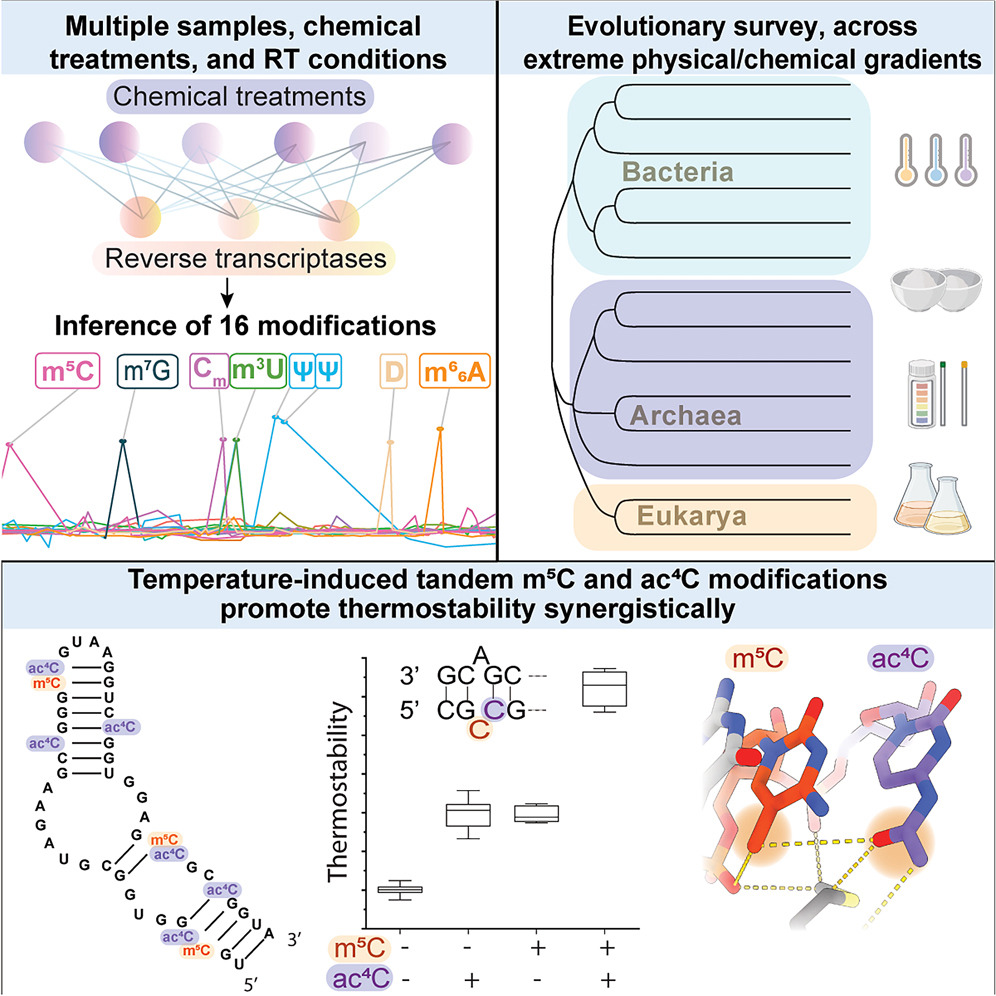
为了同时在多样本中系统检测多种rRNA修饰,作者开发了Pan-Mod-seq,其技术优势在于集成多种化学与酶学处理方法足以覆盖常见rRNA修饰;引入样本特异性条形码,支持多样本同时处理;结合多种逆转录酶,增强修饰检测的灵敏度和特异性。与此同时,团队开发称为PANORAMA的分析方法,通过线性模型对修饰数据进行定量和分类。已知修饰图谱的训练和验证显示PANORAMA具有高灵敏度和特异性。
随后,作者使用Pan-Mod-seq技术在细菌(6)、古细菌(6)和真核细胞(2)中选择14个物种在34种生长环境(温度、盐分、生长密度和pH等)中获得88个样本的rRNA修饰图谱。PANORAMA分析发现不同物种间修饰数量显著不同。具体来说,不同于大多数细菌和古细菌有20-30个修饰位点,3种物种的修饰位点明显增多:T. kodakarensis(293),P. abyssi(480)和P. furiosus(458)。值得注意的是,这3种物种的最佳生长温度为85-105℃。总之,这种修饰数量与生长温度而非系统发育的强相关表明rRNA修饰数量与生物热适应性密切相关。动态分析表明46%的修饰位点存在动态变化,尤其是假尿苷(Ψ)、5-甲基胞苷(m⁵C)和N⁴-乙酰胞苷(ac⁴C)随温度诱导上升。
紧接着,作者探究Ψ、m⁵C和ac⁴C的调节机制。对于假尿苷化,PANORAMA在T. kodakarensis鉴定到22个可能的Ψ位点,其中11个(50%)随温度升高显著诱导。由于古细菌rRNA的假尿苷化由H/ACA盒sRNAs介导,作者筛选到Tko105在65-95℃表达量升高5倍,敲除实验证实Tko105诱导至少6个位点的假尿苷化。功能学实验发现Tko105敲除株在65-75℃正常生长,但在85-95℃生长迟缓甚至缺陷,提示Tko105介导的假尿苷化改变促进热适应;对于m⁵C,PANORAMA在T. kodakarensis、P. abyssi和P. furiosus分别鉴定到22、39和94个m⁵C修饰位点。在P. furiosus,39个位点在78-102℃的m⁵C修饰增加4倍,且85%存在于GCCG基序。团队随后找到RsmB是P. furiosus负责GCCG的甲基转移酶,RsmB敲除显著抑制P. furiosus在高温度环境中的生存,提示RsmB介导m5C促进热适应。有意思的是,高温促进m⁵C既依赖RsmB酶活性的激活,也依赖高温对rRNA结构的改变;对于ac4C,rRNA乙酰化修饰主要发生在CCG基序,TkNat10作为乙酰化酶介导。类似地,高温促进ac4C同样依赖温度对TkNat10的酶活性激活和rRNA结构改变。
值得注意的是,由于修饰位点具有重叠序列,作者发现m⁵C和ac⁴C修饰常常共同发生,形成G-m⁵C-ac⁴C-G串联模块。在P. furiosus和P. abyssi中,分别有46(总48)个和17(总17)个存在m⁵C修饰的GCCG序列具有ac⁴C修饰。作者后续发现虽然GCCG的ac⁴C修饰并不是m⁵C修饰所必须的,但ac⁴C可以促进甲基化的发生。此外,单独m⁵C修饰使得Tm值增加2℃,单独ac⁴C修饰则使得Tm值增加1.9℃,而m⁵C结合ac⁴C使得Tm值增加5.2℃,提示m⁵C-ac⁴C修饰协同促进rRNA的热稳定性。冷冻电镜分析证实P. furiosus的核糖体内存在G-m⁵C-ac⁴C-G串联模块,这些甲基和乙酰基团可以通过C-H-O弱氢键与周围蛋白质和水分子形成稳定网络、填补结构空隙,共同增强极端环境的整体稳定性。
综上所述,这项研究通过Pan-Mod-seq、PANORAMA和电镜分析等技术,首次系统揭示rRNA修饰的动态性与环境适应性,为后续进一步理解rRNA表观遗传组在健康和疾病状态中的作用铺平道路。
原文链接:
https://doi.org/10.1016/j.cell.2025.09.014
1. Katherine E, Sloan., Ahmed S, Warda., Sunny, Sharma., Karl-Dieter, Entian., Denis L J, Lafontaine., Markus T, Bohnsack.(2016). Tuning the ribosome: The influence of rRNA modification on eukaryotic ribosome biogenesis and function. RNA Biol, 14(9), 1138-1152. doi:10.1080/15476286.2016.1259781
2. Sunny, Sharma., Denis L J, Lafontaine.(2015). 'View From A Bridge': A New Perspective on Eukaryotic rRNA Base Modification. Trends Biochem Sci, 40(10), 560-575. doi:10.1016/j.tibs.2015.07.008
3. Masato, Taoka., Yuko, Nobe., Yuka, Yamaki., Ko, Sato., Hideaki, Ishikawa., Keiichi, Izumikawa., Yoshio, Yamauchi., Kouji, Hirota., Hiroshi, Nakayama., Nobuhiro, Takahashi., Toshiaki, Isobe.(2018). Landscape of the complete RNA chemical modifications in the human 80S ribosome. Nucleic Acids Res, 46(18), 9289-9298. doi:10.1093/nar/gky811
4. Aldema, Sas-Chen., Justin M, Thomas., Donna, Matzov., Masato, Taoka., Kellie D, Nance., Ronit, Nir., Keri M, Bryson., Ran, Shachar., Geraldy L S, Liman., Brett W, Burkhart., et al.(2020). Dynamic RNA acetylation revealed by quantitative cross-evolutionary mapping. Nature, 583(7817), 638-643. doi:10.1038/s41586-020-2418-2
5. Aldema, Sas-Chen., Justin M, Thomas., Donna, Matzov., Masato, Taoka., Kellie D, Nance., Ronit, Nir., Keri M, Bryson., Ran, Shachar., Geraldy L S, Liman., Brett W, Burkhart., et al.(2020). Dynamic RNA acetylation revealed by quantitative cross-evolutionary mapping. Nature, 583(7817), 638-643. doi:10.1038/s41586-020-2418-2
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


